概要
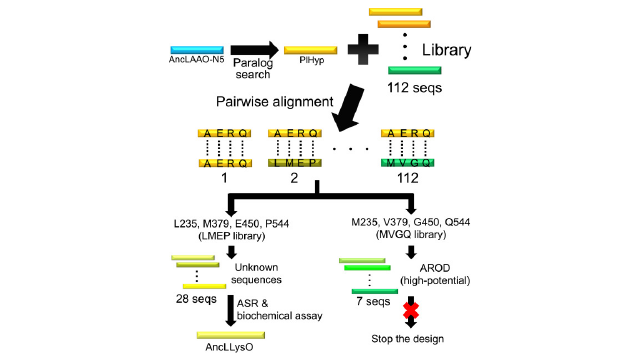
PubMedなどの公開データベースには、数多くのタンパク質の配列が登録されている。これらのデータベースには、機能的に未解明な酵素も含まれており、その中には工業的な応用が期待できるものもある。しかし、これらの酵素を割り当てることは、これまで困難な課題でした。本研究では、Chryseobacterium、Flavobacterium、Pedobactorを含むいくつかの細菌種で発現するFAD依存性酸化酵素ファミリーの未同定酵素に、合計28個のオリジナル配列を割り当てた。祖先の配列を再構成することにより、28個の配列のうち、L-リジンオキシダーゼ活性を示す子孫の配列を作成し、この酵素をAncLLysOと命名した。AncLLysOのリガンドフリー型とリガンド結合型の結晶構造を決定したところ、この酵素はR76とE383との水素結合形成によってL-Lysを認識していることがわかった。AncLLysOにL-Lysが結合すると、残基251から254で形成されるプラグループに動的な構造変化が生じることがわかった。AncLLysOの変異体を用いた生化学的解析により、これらの基質認識残基とplug loopの機能的重要性が明らかになった。R76AおよびE383D変異体も活性を失っており、G251PおよびY253A変異体のkcat/Km値は、基質が変異した残基と間接的に相互作用しているにもかかわらず、AncLLysOのそれよりも約800~1800倍も低かった。以上のことから、データベースからの配列分類と祖先の配列再構築を組み合わせたアプローチは、未知の配列のデータベースを用いて新しい酵素を見つけるだけでなく、その機能を解明するためにも有効であることが示された。